J. de Ridder et ses collègues ont mené une étude sur le développement et l’utilisation d’un classificateur à réseau neuronal appelé Sturgeon pour classer les tumeurs du système nerveux central (SNC) pendant la chirurgie. Ils ont récemment publié leurs travaux dans Nature (DOI:10.1038/s41586-023-06615-2). L’équipe a abordé les limites des méthodes actuelles pour déterminer les types de tumeurs précis avant la chirurgie et a exploré les avantages potentiels de l’utilisation du séquençage rapide de nanopores pour obtenir un profil de méthylation pendant la chirurgie. Le classificateur Sturgeon, optimisé pour gérer les données rares, a été entraîné sur des simulations de séquençage de nanopores. L’étude a démontré que Sturgeon pouvait classer avec précision les échantillons de tumeurs du SNC sur la base d’aussi peu que 20-40 minutes de temps de séquençage. Dans un contexte intraopératoire réaliste, Sturgeon a démontré sa capacité à influencer la prise de décision chirurgicale en fournissant un temps de rotation diagnostique de moins de 90 minutes. L’importance d’un diagnostic précis et fiable pour guider les stratégies neurochirurgicales a été soulignée. L’utilisation du séquençage de nanopores a offert des avantages tels que le faible coût de mise en place, la petite taille de forme, et la disponibilité immédiate des données. Bien que l’approche Sturgeon nécessite une certaine quantité de tissu, des tests réussis avec de plus petits échantillons ont suggéré qu’elle pourrait aider à prévenir la comorbidité neurologique et la nécessité de chirurgies supplémentaires.
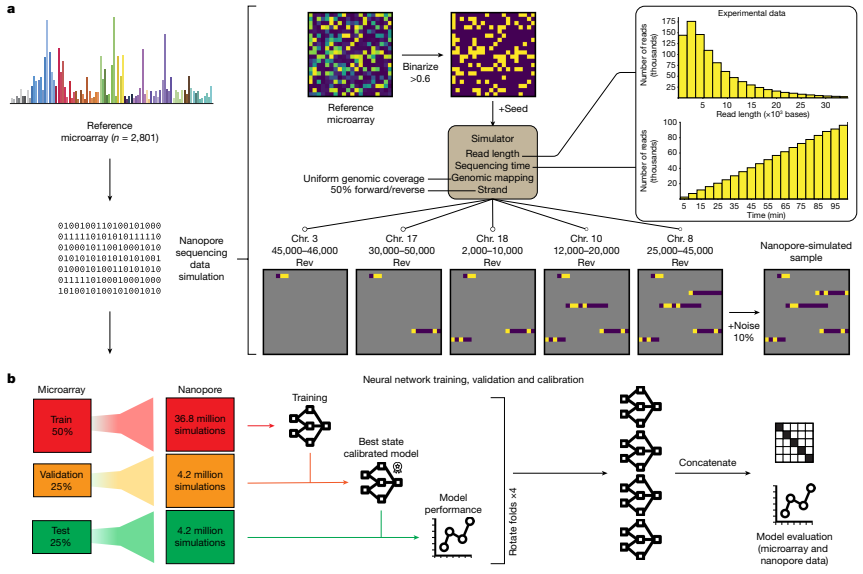
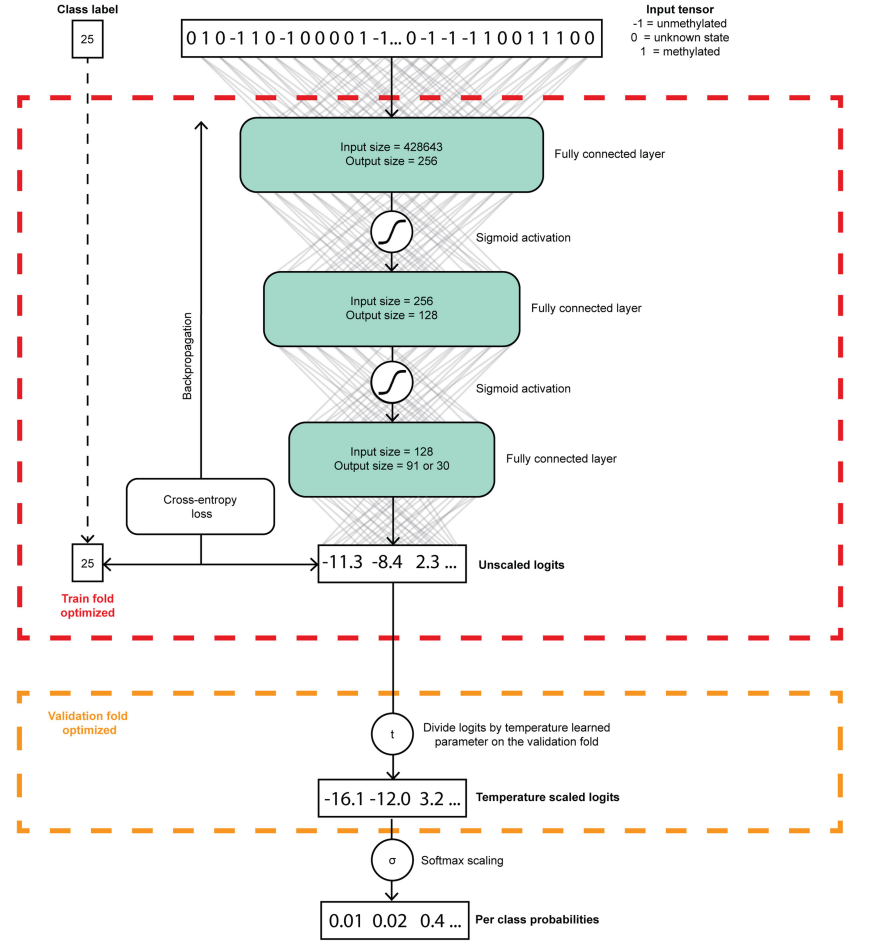
Les chercheurs ont utilisé des données simulées de séquençage de nanopores pour entraîner et vérifier leur modèle Sturgeon pour la classification des tumeurs du SNC. Plus précisément, ils ont utilisé quatre réseaux neuronaux qui ont été chacun entraînés, validés, et calibrés indépendamment. Les données d’entraînement étaient constituées de simulations générées à partir de l’ensemble de référence de Capper et al., qui a été divisé en quatre plis tout en conservant les distributions de classe originales. Les chercheurs ont utilisé two plis pour entraîner le sous-modèle, one pli pour déterminer l’état le plus performant du sous-modèle et effectuer le calibrage du score, et le dernier pli pour évaluer les performances du sous-modèle. Les réseaux neuronaux ont été pré-entraînés en utilisant des simulations qui variaient de 0,6% à 14% de rareté, puis affinés pour le classificateur final en utilisant des simulations qui variaient de 0,6% à 6,3% de rareté. Pour vérifier les performances du modèle, les chercheurs ont évalué sa performance finale sur un pli de test laissé de côté, chaque échantillon contribuant 6 000 échantillons simulés à l’ensemble de test. Ils ont évalué la précision de la classification et d’autres mesures sur tous les niveaux de rareté pour évaluer les performances du modèle. Les chercheurs ont également validé leur modèle sur des profils de méthylation pédiatriques obtenus à partir de processus diagnostiques de routine. De plus, ils ont effectué la classification de données de séquençage de nanopores disponibles au public obtenues à partir de GSE209865. En bref, le modèle Sturgeon a été formé et vérifié à l’aide de données de séquençage de nanopores simulées, y compris des simulations de l’ensemble de référence de Capper et al. et des données de séquençage de nanopores disponibles au public. Les performances du modèle ont été évaluées à l’aide de différentes mesures et validées sur de véritables profils de méthylation pédiatriques.
L’ensemble de données d’entraînement pour Sturgeon se compose d’une population variée de patients de différents âges (âge moyen de 29 ans avec 36% de moins de 13 ans). Notre premier objectif était de valider davantage les performances de Sturgeon dans un contexte pédiatrique. À cette fin, nous avons obtenu 94 profils de méthylation génomique réalisés à l’aide des tableaux Illumina Infinium Methyla-tionEPIC v1.0 (désignés ci-après par EPIC) de patients ayant subi une chirurgie de résection d’une tumeur du SNC dans le Centre Princess Máxima (PMC) pour l’oncologie pédiatrique. Pour chacun de ces échantillons, le classificateur de Heidelberg disponible au public (v11b4) a été appliqué comme partie de la prise en charge clinique de routine. Ce classificateur peut être considéré comme une version mise à jour du classificateur de Capper et al. La coupure recommandée pour un diagnostic clinique est de 0,84, que le classificateur a atteint pour la majorité (n = 68) des échantillons. Ceux classifiés en dessous de la coupure de 0,84 sont considérés comme difficiles à diagnostiquer sur la base de leur profil de méthylation, ce qui est susceptible de se produire pour des types de tumeurs peu communs qui ne correspondent à aucun des classes précédemment annotées, des tumeurs qui surviennent dans le contexte d’un syndrome de prédisposition tumorale génétique, des échantillons hétérogènes ou des échantillons ayant une faible pureté tumorale.Pour chaque profil de méthylation, nous avons simulé 500 expériences de séquençage de nanopores à 7 profondeurs de séquençage pour un total de 332 500 expériences de séquençage de nanopores simulées, après quoi nous avons appliqué le classificateur Sturgeon
Les neurochirurgiens peuvent utiliser le classificateur de réseau neuronal Sturgeon pendant la chirurgie pour prédire la classification des tumeurs. Le classificateur Sturgeon est basé sur le séquençage rapide de nanopores, ce qui permet l’acquisition d’un profil de méthylation de la tumeur pendant la procédure chirurgicale [1]. Cette méthode surmonte les limites de l’imagerie pré-opératoire actuelle et de l’analyse histologique peropératoire, qui ne peuvent pas toujours fournir un diagnostic de type tumoral concluant ou précis.
Le classificateur Sturgeon est optimisé pour gérer les données rares et a été entraîné sur des courses de séquençage de nanopores simulées [2]. Il peut classer précisément les échantillons de tumeurs du SNC basés sur aussi peu que 20-40 minutes de séquençage [2]. Dans un contexte intraopératoire réaliste, Sturgeon a démontré sa capacité à fournir un temps de rotation diagnostique de moins de 90 minutes, influençant la prise de décision chirurgicale [1]. En obtenant rapidement un diagnostic de sous-type moléculaire, les neurochirurgiens peuvent prendre des décisions plus éclairées quant à l’étendue de la résection de la tumeur, minimisant ainsi le risque de dommages neurologiques et de comorbidité.
L’utilisation du séquençage de nanopores dans l’approche Sturgeon offre des avantages tels que le faible coût de mise en place, un petit facteur de forme, et la disponibilité instantanée des données. Bien qu’une certaine quantité de tissu soit actuellement nécessaire pour l’approche Sturgeon, elle a été testée avec succès sur des échantillons plus petits. En somme, l’utilisation du classificateur Sturgeon pendant la chirurgie peut aider les neurochirurgiens à obtenir une classification précise et fiable des tumeurs, guidant leurs stratégies chirurgicales et potentiellement en prévenant la morbidité neurologique et le besoin de chirurgies supplémentaires.